Plant Biotechnology Journal |我院李自超教授团队张洪亮/张战营发现参与水稻分蘖建成的自然变异新基因
水稻的分蘖数是决定有效穗数的一个关键因素,其对水稻的最终产量有着较为直接的影响。近日,中国农业大学农学院李自超教授团队张洪亮/张战营合作研究克隆了一个新的水稻分蘖形成调控基因,并对其分子作用机制和潜在的育种利用价值进行了深入分析。相关研究结果以research article形式在线发表在植物学知名期刊 《Plant Biotechnology Journal》上(IF=13.263)。
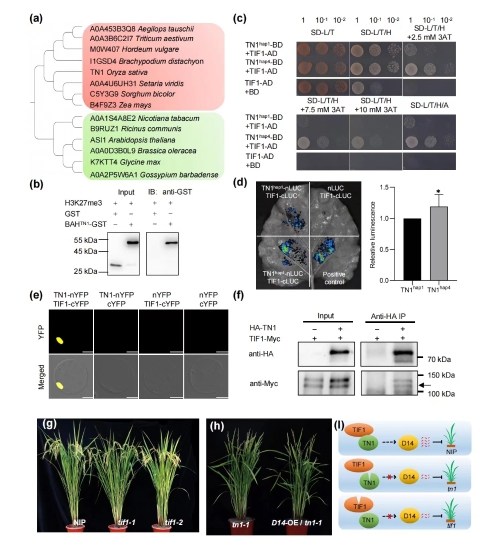
该研究团队利用295份来源广泛的水稻种质材料进行全基因组穗数关联分析,在1号染色体鉴定到一个显著关联的QTL,结合单倍型分析和种质材料表达分析,在该区间内确定了一个候选基因Tiller Number1(TN1),其编码一个含有BAH结构域和RNA识别结构域的功能蛋白,其BAH结构域具有H3K27me3组蛋白修饰识别功能。转基因表型评价发现,TN1负调控水稻分蘖数及穗数的建成。拟南芥中TN1的同源蛋白AtASI1,能够与AIPP1和EDM2互作形成AAE复合体,调控下游靶基因的表达(Duan et al., 2017)。本研究中作者克隆了AIPP1在水稻中的同源基因TIF1。与TN1基因敲除植株表型类似,TIF1基因敲除植株分蘖数及穗数显著增加。进一步研究发现位于TN1基因BAH结构域G→A(+2163)的自然变异,能够影响TN1与TIF1的互作强度。
在植物体内AAE复合体参与了多种转录和转录后调节途径。为了进一步探究TN1可能的下游基因,该研究通过转录组测序和荧光定量表达分析发现TN1和TIF1功能缺失后,均会造成水稻分蘖调控关键基因D3、D14、OsCCA1的表达下调。前人报道NGR5能够影响D14基因组的H3K27me3修饰水平来调控D14表达(Wu et al., 2020)。而TN1具有H3K27me3识别功能,说明TN1可能通过介导D14基因区H3K27me3修饰丰度变化,进而影响D14的表达。D14在tn1-1突变体中的过量表达能够显著减少分蘖,进一步从遗传角度说明TN1-TIF1通过调控D14的表达进而影响水稻分蘖建成。
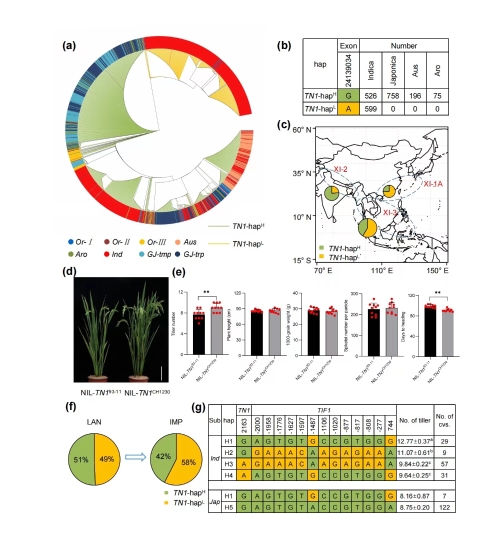
为了进一步探究TN1自然变异的潜在育种价值,作者结合水稻3K测序数据和野生稻测序数据(Huang et al., 2012;Wang et al., 2018)进一步分析发现含有TN1-hapH等位基因的材料具有较多分蘖,广泛存在于各个稻种亚群,主要分布在东亚和东南亚地区;而含有TN1-hapL等位基因的材料分蘖较少,仅存在于籼稻亚群,主要分布在南亚地区。作者进一步以93-11品种(TN1-hapL等位基因)为受体亲本,CH1230(TN1-hapH等位基因)为供体亲本构建近等基因系。结果表明NIL-TN1CH1230的分蘖数和穗数显著高于NIL-TN193-11,说明TN1-hapH等位基因对水稻分蘖数具有增效作用。联合单倍型分析表明,聚合TN1和TIF1的优异等位基因,可将有效改良部分籼稻品种的分蘖及穗数,具有较大的育种利用潜力。
综上,该研究鉴定了一个新的水稻分蘖形成调控自然变异TN1,并明确了其参与水稻分蘖建成的“TN1-TIF1-D14”分子调控模块,评估了种质资源中不同TN1基因型的育种利用价值,为水稻株型及产量的分子遗传改良提供了优异基因资源和遗传材料。
中国农业大学农学院博士生张全和已出站博士后谢建引为论文第一作者,中国农业大学农学院张战营副教授和中国农业大学农学院/海南大学三亚南繁研究院/三亚中国农业大学研究院张洪亮教授为论文的通讯作者。中国农业大学农学院李自超教授、李金杰副教授,广西农科院水稻研究所李丹婷研究员、潘英华副研究员和梁云涛研究员参与了指导。该研究得到了国家自然科学基金面上项目、十三五国家重点研发计划、三亚崖州湾科技城项目和广西农科院开放课题等项目的资助。
图1. TN1候选基因分析及功能验证
图2. “TN1-TIF1-D14”分子调控模块参与水稻分蘖建成
图3. TN1自然变异的育种价值分析
原文链接:https://onlinelibrary.wiley.com/doi/10.1111/pbi.14017
参考文献:
Duan, C., Wang, X., Zhang, L., Xiong, X., Zhang, Z., Tang, K., et al. (2017) A protein complex regulates RNA processing of intronic heterochromatin-containing genes in Arabidopsis. Proc. Natl. Acad. Sci. USA 114, E7377-E7384.
Huang, X., Kurata, N., Wei, X., Wang, Z.-X., Wang, A., Zhao, Q., Zhao, Y. et al. (2012) A map of rice genome variation reveals the origin of cultivated rice. Nature 490, 497-501.
Wang, W., Mauleon, R., Hu, Z., Chebotarov, D., Tai, S., Wu, Z., Li, M. et al. (2018) Genomic variation in 3,010 diverse accessions of Asian cultivated rice. Nature 557, 43-49.
Wu, K., Wang, S., Song, W., Zhang, J., Wang, Y., Liu, Q., et al. (2020) Enhanced sustainable green revolution yield via nitrogen-responsive chromatin modulation in rice. Science 367, eaaz2046.